Введение.
Python замечательный язык. Связка Python + NumPy + Matplotlib, на мой взгляд, сейчас одна из лучших для научных расчётов и быстрого прототипирования алгоритмов. Но у каждого инструмента есть свои светлые и тёмные стороны. Одной из самых дискутируемых особенностей Python является GIL – Global Interpreter Lock. Я бы отнёс эту особенность к тёмной стороне инструмента. Хотя многие со мной не согласятся.
Если кратко, то GIL не позволяет в одном интерпретаторе Python эффективно использовать больше одного потока. Защитники GIL утверждают, что однопоточные программы при наличии GIL работают намного эффективнее. Но наличие GIL означает, что параллельные вычисления с использованием множества потоков и общей памяти невозможны. А это достаточно сильное ограничения в современном многоядерном мире.
Один из способов преодоления GIL при помощи потоков на C++ был недавно рассмотрен в статье: Использование Python в многопоточном приложении на C++. Я же хочу рассмотреть другой способ преодоления ограничений GIL, основанный на multiprocessing и shared array. На мой взгляд, этот способ позволяет достаточно просто и эффективно использовать процессы и разделяемую память для прозрачного параллельного программирования в стиле множества потоков и общей памяти.
Задача.
В качестве примера рассмотрим следующую задачу. В трёхмерном пространстве заданы N точек v0, v1, …, vN. Требуется для каждой пары точек вычислить функцию, зависящую от расстояния между ними. Результат будет представлять собой матрицу NxN со значениями этой функции. В качестве функции возьмем следующую: f = r^3 / 12 + r^2 / 6. Этот тест, на самом деле, не такой уж и синтетический. На вычислении таких функций от расстояния основана RBF интерполяция, которая используется во многих областях вычислительной математики.
Способ параллелизации.
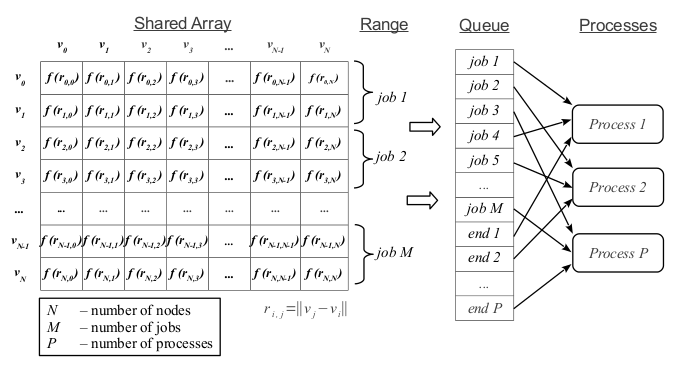
В данной задаче каждая строка матрицы может вычисляться независимо. Из каждых нескольких строк матрицы сформируем независимые работы и поместим их в очередь заданий (см. рис. 1). Запустим несколько процессов. Каждый процесс будет брать из очереди следующее задание на выполнение, пока не встретит специальное задание с кодом «end». В этом случае процесс заканчивает свою работу.
Реализация на Python.
В реализации на Python у нас будут два главных метода: mpCalcDistance(nodes) и
mpCalcDistance_Worker(nodes, queue, arrD). Метод mpCalcDistance(nodes) принимает на вход список узлов, создает область общей памяти, подготавливает очередь заданий и запускает процессы. Метод mpCalcDistance_Worker(nodes, queue, arrD) это вычислительный метод, работающий в собственном потоке. Он принимает на вход список узлов, очередь заданий и область общей памяти. Рассмотрим реализацию этих методов подробнее.
Метод mpCalcDistance(nodes)
Создаем область общей памяти:
nP = nodes.shape[0]
nQ = nodes.shape[0]
arrD = mp.RawArray(ctypes.c_double, nP * nQ)
Создаем очередь заданий. Каждое задание не что иное, как просто диапазон строчек матрицы. Специальное значение None это признак завершения работы процесса:
nCPU = 2
nJobs = nCPU * 36
q = nP / nJobs
r = nP % nJobs
jobs = []
firstRow = 0
for i in range(nJobs):
rowsInJob = q
if (r > 0):
rowsInJob += 1
r -= 1
jobs.append((firstRow, rowsInJob))
firstRow += rowsInJob
queue = mp.JoinableQueue()
for job in jobs:
queue.put(job)
for i in range(nCPU):
queue.put(None)
Создаем процессы и ждем пока не закончится очередь заданий:
workers = []
for i in range(nCPU):
worker = mp.Process(target = mpCalcDistance_Worker,
args = (nodes, queue, arrD))
workers.append(worker)
worker.start()
queue.join()
Из области общей памяти формируем матрицу с результатами:
D = np.reshape(np.frombuffer(arrD), (nP, nQ))
return D
Метод mpCalcDistance_Worker(nodes, queue, arrD)
Оборачиваем область общей памяти в матрицу:
nP = nodes.shape[0]
nQ = nodes.shape[0]
D = np.reshape(np.frombuffer(arrD), (nP, nQ))
Пока не закончилась очередь заданий берем следующее задание из очереди и выполняем расчет:
while True:
job = queue.get()
if job == None:
break
start = job[0]
stop = job[0] + job[1]
# components of the distance vector
p = nodes[start:stop]
q = nodes.T
Rx = p[:, 0:1] - q[0:1]
Ry = p[:, 1:2] - q[1:2]
Rz = p[:, 2:3] - q[2:3]
# calculate function of the distance
L = np.sqrt(Rx * Rx + Ry * Ry + Rz * Rz)
D[start:stop, :] = L * L * L / 12 + L * L / 6
queue.task_done()
Результаты
Среда выполнения: двухядерный процессор, Ubuntu 12.04, 64bit.
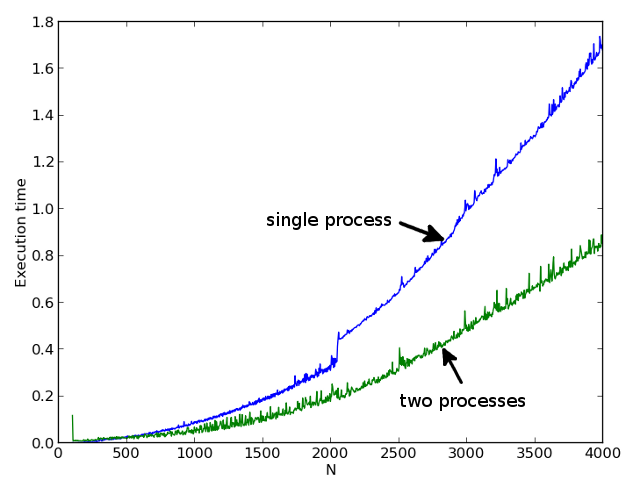
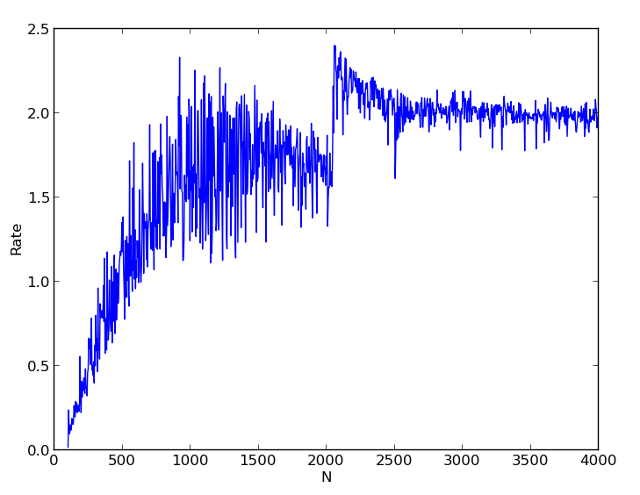
На первой картинке показаны времена однопоточного и двухпоточного расчета для различных N. На второй картинке показано отношение времени однопоточного расчета к двухпоточному. Видно, что начиная с N = 500 мы получаем уже существенное ускорение расчета.
Очень интересный результат получается в районе числа N = 2000. В однопоточном варианте мы получаем заметный скачок времени расчета, а при многопоточном расчете коэффициент ускорения даже превышает 2. Я объясняю это эффектом кэша. В многопоточном варианте данные для каждой задачи полностью умещаются в кэш. А в однопоточном уже нет.
Так что использование процессов и общей памяти, на мой взгляд, достаточно просто позволяет обойти ограничения GIL.
Полный код всего скрипта:
""" Python multiprocessing with shared memory example.
This example demonstrate workaround for the GIL problem. Workaround uses
processes instead of threads and RawArray allocated from shared memory.
See also:
[1] http://docs.python.org/2/library/multiprocessing.html
[2] http://folk.uio.no/sturlamo/python/multiprocessing-tutorial.pdf
[3] http://www.bryceboe.com/2011/01/28/the-python-multiprocessing-queue-and-large-objects/
"""
import time
import ctypes
import multiprocessing as mp
import numpy as np
import matplotlib.pyplot as plt
def generateNodes(N):
""" Generate random 3D nodes
"""
return np.random.rand(N, 3)
def spCalcDistance(nodes):
""" Single process calculation of the distance function.
"""
p = nodes
q = nodes.T
# components of the distance vector
Rx = p[:, 0:1] - q[0:1]
Ry = p[:, 1:2] - q[1:2]
Rz = p[:, 2:3] - q[2:3]
# calculate function of the distance
L = np.sqrt(Rx * Rx + Ry * Ry + Rz * Rz)
D = L * L * L / 12 + L * L / 6
return D
def mpCalcDistance_Worker(nodes, queue, arrD):
""" Worker process for the multiprocessing calculations
"""
nP = nodes.shape[0]
nQ = nodes.shape[0]
D = np.reshape(np.frombuffer(arrD), (nP, nQ))
while True:
job = queue.get()
if job == None:
break
start = job[0]
stop = job[0] + job[1]
# components of the distance vector
p = nodes[start:stop]
q = nodes.T
Rx = p[:, 0:1] - q[0:1]
Ry = p[:, 1:2] - q[1:2]
Rz = p[:, 2:3] - q[2:3]
# calculate function of the distance
L = np.sqrt(Rx * Rx + Ry * Ry + Rz * Rz)
D[start:stop, :] = L * L * L / 12 + L * L / 6
queue.task_done()
queue.task_done()
def mpCalcDistance(nodes):
""" Multiple processes calculation of the distance function.
"""
# allocate shared array
nP = nodes.shape[0]
nQ = nodes.shape[0]
arrD = mp.RawArray(ctypes.c_double, nP * nQ)
# setup jobs
#nCPU = mp.cpu_count()
nCPU = 2
nJobs = nCPU * 36
q = nP / nJobs
r = nP % nJobs
jobs = []
firstRow = 0
for i in range(nJobs):
rowsInJob = q
if (r > 0):
rowsInJob += 1
r -= 1
jobs.append((firstRow, rowsInJob))
firstRow += rowsInJob
queue = mp.JoinableQueue()
for job in jobs:
queue.put(job)
for i in range(nCPU):
queue.put(None)
# run workers
workers = []
for i in range(nCPU):
worker = mp.Process(target = mpCalcDistance_Worker,
args = (nodes, queue, arrD))
workers.append(worker)
worker.start()
queue.join()
# make array from shared memory
D = np.reshape(np.frombuffer(arrD), (nP, nQ))
return D
def compareTimes():
""" Compare execution time single processing versus multiple processing.
"""
nodes = generateNodes(3000)
t0 = time.time()
spD = spCalcDistance(nodes)
t1 = time.time()
print "single process time: {:.3f} s.".format(t1 - t0)
t0 = time.time()
mpD = mpCalcDistance(nodes)
t1 = time.time()
print "multiple processes time: {:.3f} s.".format(t1 - t0)
err = np.linalg.norm(mpD - spD)
print "calculate error: {:.2e}".format(err)
def showTimePlot():
""" Generate execution time plot single processing versus multiple processing.
"""
N = range(100, 4000, 4)
spTimes = []
mpTimes = []
rates = []
for i in N:
print i
nodes = generateNodes(i)
t0 = time.time()
spD = spCalcDistance(nodes)
t1 = time.time()
sp_tt = t1 - t0
spTimes.append(sp_tt)
t0 = time.time()
mpD = mpCalcDistance(nodes)
t1 = time.time()
mp_tt = t1 - t0
mpTimes.append(mp_tt)
rates.append(sp_tt / mp_tt)
plt.figure()
plt.plot(N, spTimes)
plt.plot(N, mpTimes)
plt.xlabel("N")
plt.ylabel("Execution time")
plt.figure()
plt.plot(N, rates)
plt.xlabel("N")
plt.ylabel("Rate")
plt.show()
def main():
compareTimes()
#showTimePlot()
if __name__ == '__main__':
main()